Table of contents
本ファイルは,chatgpt支援のもと,シミュレーションデータを用いてエントロピーバランシングを実装してみる.
エントロピーバランシングの定式化
エントロピーを最大化するようなwを見つけたい
ここでいうエントロピーとは情報理論におけるエントロピー,情報エントロピー のことであり,確率変数が持つ情報の量をさす.
エントロピーは以下の式で与えられる \[
H(w) = -\sum_{i = 1}^{n_0}w_i \log w_i
\] 左辺のエントロピーを最大化するような確率変数wは,右辺を最小化するような確率変数wということである.よって,以下の目的関数を考えれば良い.
\[
\min_w \sum_{i = 1}^{n_0}w_i \log w_i
\]
エントロピーを最大化するようなwに対して,3つ制約を課す
おそらく,本命はモーメント一致であり,この制約を満たす確率変数wののうちから,エントロピーが最大なwを採用する,といった感じだろうか.最大エントロピー原理によって正当化される気がする.つまり,制約のもとで最もエントロピー(=不確実性が高い)ものを採用することは,制約以外の異物,エコノメ風にいうならバイアス,が入ってないので,そのような確率変数wが最も妥当である.
ebal packageによる実装
##
## ebal Package: Implements Entropy Balancing.
## See http://www.stanford.edu/~jhain/ for additional information.
# データ生成(再掲) set.seed (123 )<- 500 <- rnorm (n)<- rbinom (n, 1 , 0.5 )<- runif (n)<- 0.5 * X1 - 0.8 * X2 + 0.3 * X3<- 1 / (1 + exp (- logit_p))<- rbinom (n, 1 , p)<- data.frame (X1, X2, X3, treatment)# 共変量マトリクス <- as.matrix (df[, c ("X1" , "X2" , "X3" )])<- df$ treatment # 0=control, 1=treated # 実行:対照群を処置群に一致させる重みを学習 <- ebalance (Treatment = Treatment, X = X_all)
Converged within tolerance
# 検証:処置群平均 <- colMeans (X_all[Treatment == 1 , ])# 重み付き対照群平均 <- apply (X_all[Treatment == 0 , ], 2 , weighted.mean, w = eb$ w)# 比較 round (treated_mean, 3 )
X1 X2 X3
0.254 0.413 0.518
round (weighted_control_mean, 3 )
X1 X2 X3
0.254 0.413 0.518
エントロピーで重みづけることでバランシングしたようだ.バランシングしたようだというのは正しい表現ではないようだ.そもそも制約式に,wというウェイトで平均を取ると,処置群の平均に一致する問い条件がある.これこそまさにバランシングさせてるに等しい.wでバランシングするのはなぜか,という問いの答えは,バランシングするように計算されたのがwだから,である.
# アウトカムの生成(真のτ = 2) set.seed (123 )<- rnorm (n, 0 , 1 )<- 2 * treatment + 0.3 * X1 - 0.5 * X2 + 0.2 * X3 + epsilon$ Y <- Y# 処置群の平均 <- mean (df$ Y[Treatment == 1 ])# 重み付き対照群の平均 <- weighted.mean (df$ Y[Treatment == 0 ], w = eb$ w)# ATT = 処置群平均 - 重み付き対照群平均 <- Y_treated - Y_control_weightedcat ("直接計算された ATT:" , round (ATT, 3 ), " \n " )
ブートストラップによる信頼区間の構築を目指す.
# ライブラリ library (ebal)library (dplyr)
Attaching package: 'dplyr'
The following objects are masked from 'package:stats':
filter, lag
The following objects are masked from 'package:base':
intersect, setdiff, setequal, union
# 関数定義:1回のATT推定 <- function (df) {<- as.matrix (df[, c ("X1" , "X2" , "X3" )])<- df$ Y<- df$ treatmentinvisible (capture.output (<- ebalance (Treatment = treatment, X = X)<- mean (Y[treatment == 1 ])<- weighted.mean (Y[treatment == 0 ], w = eb$ w)return (treated_mean - control_weighted_mean)# ブートストラップ set.seed (42 )<- 500 <- replicate (B, {<- df[sample (1 : nrow (df), replace = TRUE ), ]tryCatch (estimate_att (df_boot), error = function (e) NA )# 結果 <- quantile (att_boot, probs = c (0.025 , 0.975 ), na.rm = TRUE )cat ("ATT 推定値のブートストラップ95%信頼区間:" , round (boot_ci, 3 ), " \n " )
ATT 推定値のブートストラップ95%信頼区間: 1.998 2
Weight packageによる実装
# --- パッケージの読み込み --- library (WeightIt)library (cobalt) # バランスチェック
cobalt (Version 4.6.0, Build Date: 2025-04-15)
Loading required package: grid
Loading required package: Matrix
Loading required package: survival
Attaching package: 'survey'
The following object is masked from 'package:WeightIt':
calibrate
The following object is masked from 'package:graphics':
dotchart
# --- データ生成(ATE = 2) --- set.seed (123 )<- 1000 <- rnorm (n)<- rbinom (n, 1 , 0.5 )<- plogis (0.5 * X1 - 0.7 * X2)<- rbinom (n, 1 , pscore)<- 1 + 0.5 * X1 - 0.3 * X2 + rnorm (n)<- y0 + 2 <- ifelse (treat == 1 , y1, y0)<- data.frame (y, treat, X1, X2)
# --- ATE, ATT, ATCごとの重み計算 --- <- weightit (treat ~ X1 + X2, data = dat, method = "ebal" , estimand = "ATE" )<- weightit (treat ~ X1 + X2, data = dat, method = "ebal" , estimand = "ATT" )<- weightit (treat ~ X1 + X2, data = dat, method = "ebal" , estimand = "ATC" )# --- 重み付きデザインの作成 --- <- svydesign (ids = ~ 1 , weights = ~ w_ate$ weights, data = dat)<- svydesign (ids = ~ 1 , weights = ~ w_att$ weights, data = dat)<- svydesign (ids = ~ 1 , weights = ~ w_atc$ weights, data = dat)# --- 回帰モデルによる推定(y ~ treat) --- <- svyglm (y ~ treat, design = des_ate)<- svyglm (y ~ treat, design = des_att)<- svyglm (y ~ treat, design = des_atc)#計算結果 cat ("ATE 推定値と信頼区間: \n " )
Call:
svyglm(formula = y ~ treat, design = des_ate)
Survey design:
svydesign(ids = ~1, weights = ~w_ate$weights, data = dat)
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 0.84065 0.04550 18.48 <2e-16 ***
treat 1.98743 0.07342 27.07 <2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for gaussian family taken to be 1.164528)
Number of Fisher Scoring iterations: 2
Call:
svyglm(formula = y ~ treat, design = des_att)
Survey design:
svydesign(ids = ~1, weights = ~w_att$weights, data = dat)
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 1.02444 0.05305 19.31 <2e-16 ***
treat 1.99661 0.07648 26.11 <2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for gaussian family taken to be 1.163522)
Number of Fisher Scoring iterations: 2
Call:
svyglm(formula = y ~ treat, design = des_atc)
Survey design:
svydesign(ids = ~1, weights = ~w_atc$weights, data = dat)
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 0.71376 0.04431 16.11 <2e-16 ***
treat 1.98070 0.07880 25.14 <2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for gaussian family taken to be 1.154199)
Number of Fisher Scoring iterations: 2
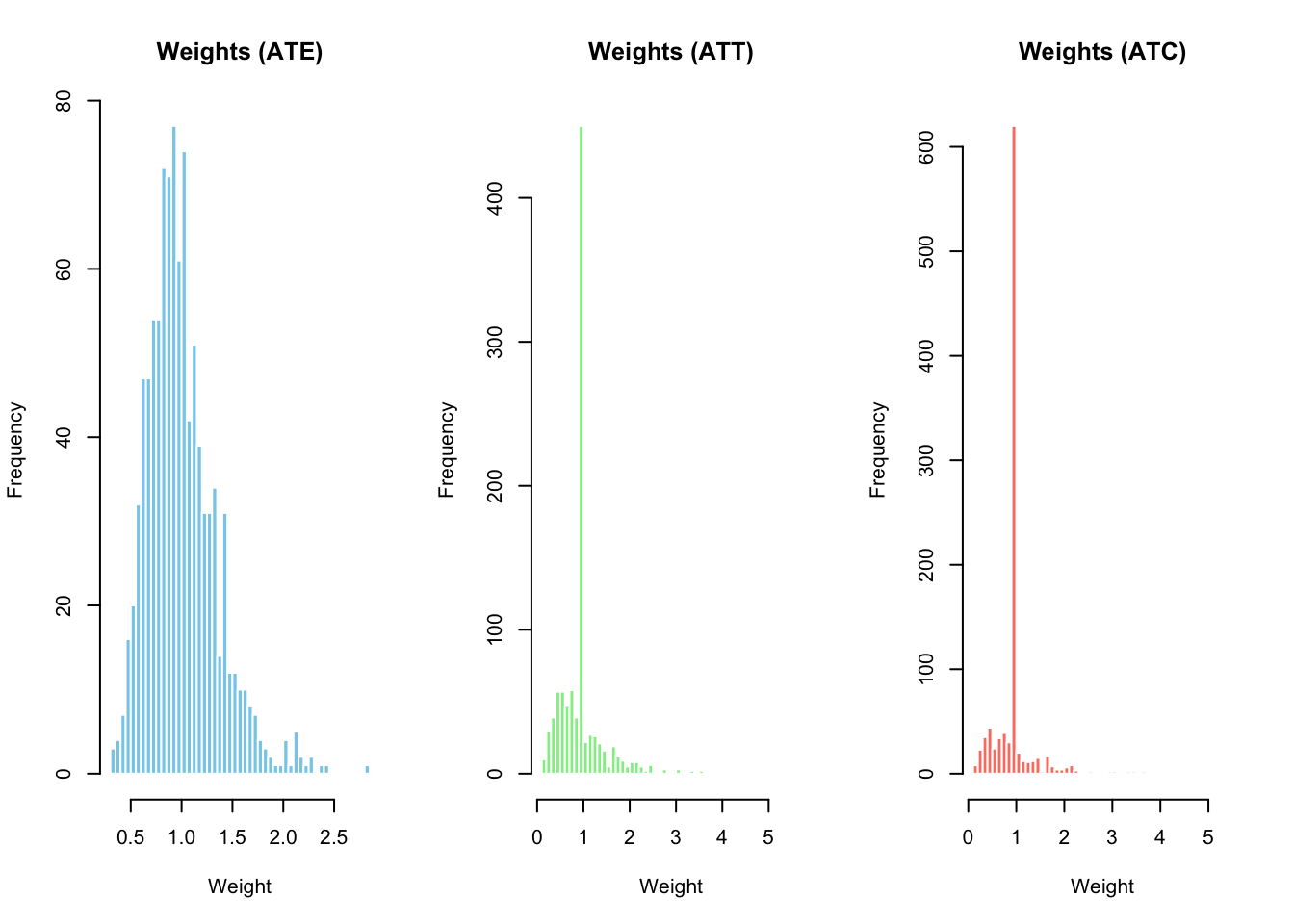
par (mfrow = c (1 , 3 )) # 1行3列に分割表示 hist (w_ate$ weights,breaks = 50 ,main = "Weights (ATE)" ,xlab = "Weight" , col = "skyblue" , border = "white" )hist (w_att$ weights,breaks = 50 ,main = "Weights (ATT)" ,xlab = "Weight" , col = "lightgreen" , border = "white" )hist (w_atc$ weights,breaks = 50 ,main = "Weights (ATC)" ,xlab = "Weight" , col = "salmon" , border = "white" )par (mfrow = c (1 , 1 )) # レイアウトを戻す
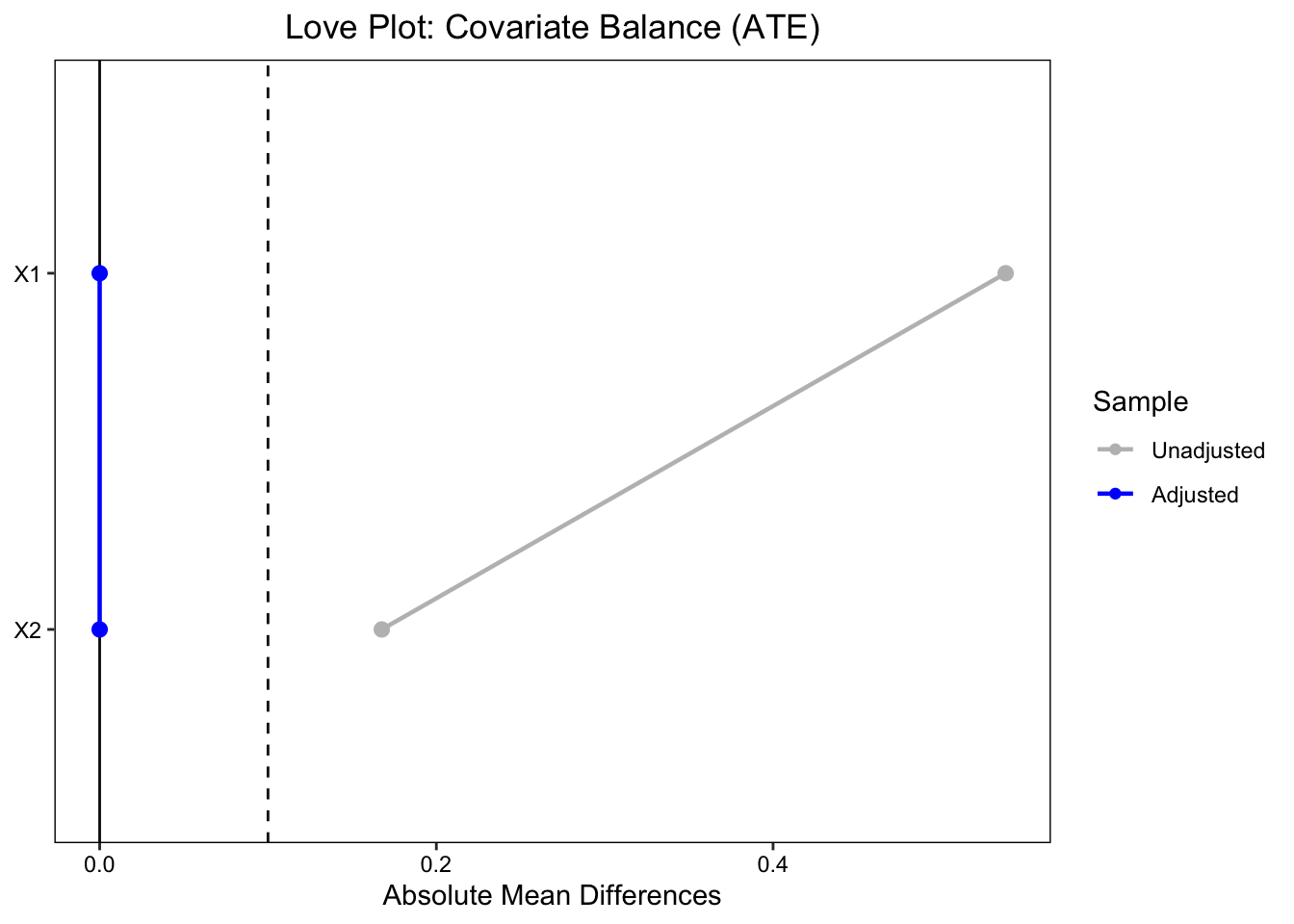
# ATE love.plot (w_ate,stat = "mean.diffs" , abs = TRUE , thresholds = c (m = .1 ),var.order = "unadjusted" , line = TRUE ,title = "Love Plot: Covariate Balance (ATE)" ,colors = c ("grey" , "blue" ))
Warning: Standardized mean differences and raw mean differences are present in
the same plot. Use the `stars` argument to distinguish between them and
appropriately label the x-axis. See `?love.plot` for details.
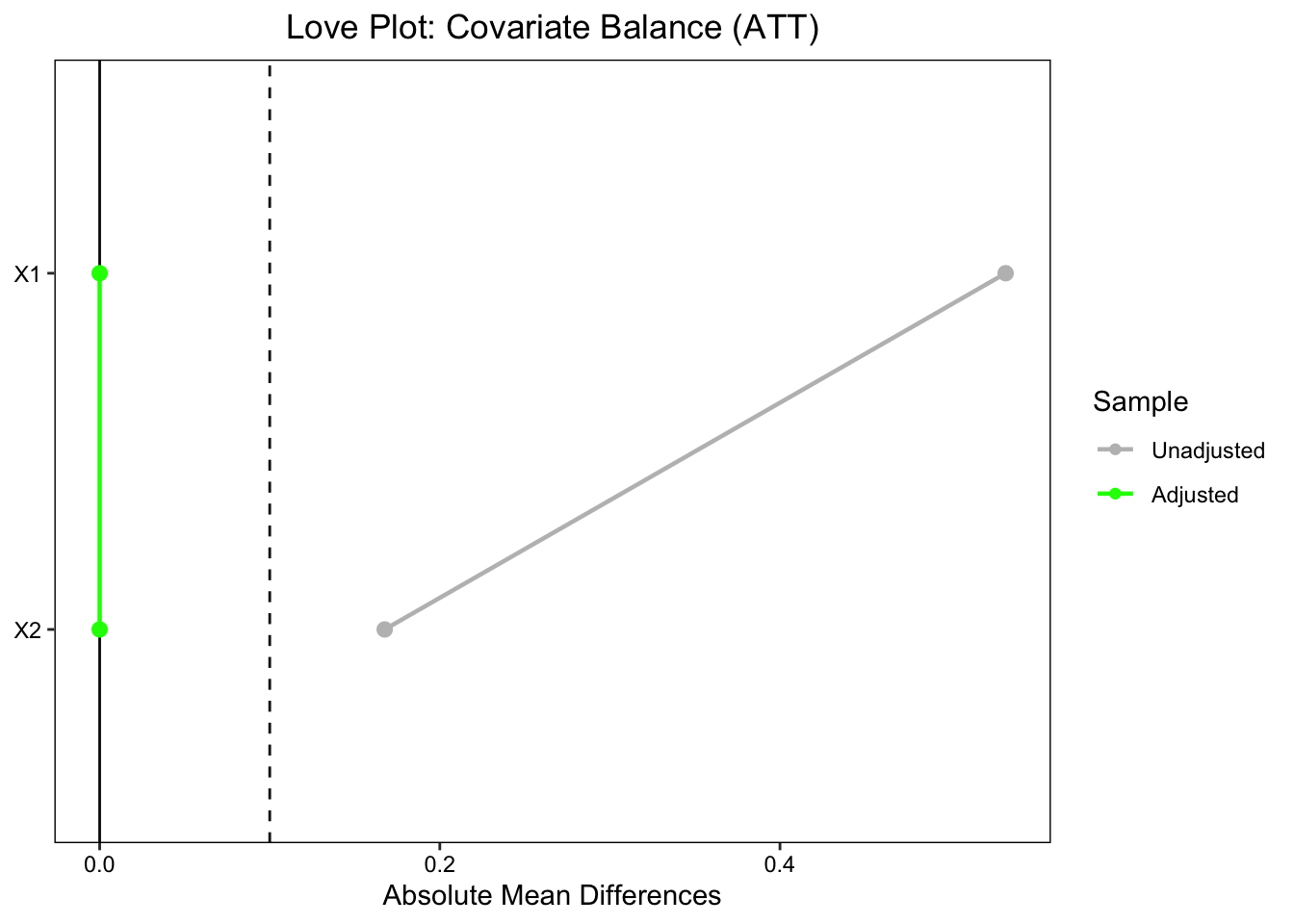
# ATT love.plot (w_att,stat = "mean.diffs" , abs = TRUE , thresholds = c (m = .1 ),var.order = "unadjusted" , line = TRUE ,title = "Love Plot: Covariate Balance (ATT)" ,colors = c ("grey" , "green" ))
Warning: Standardized mean differences and raw mean differences are present in
the same plot. Use the `stars` argument to distinguish between them and
appropriately label the x-axis. See `?love.plot` for details.
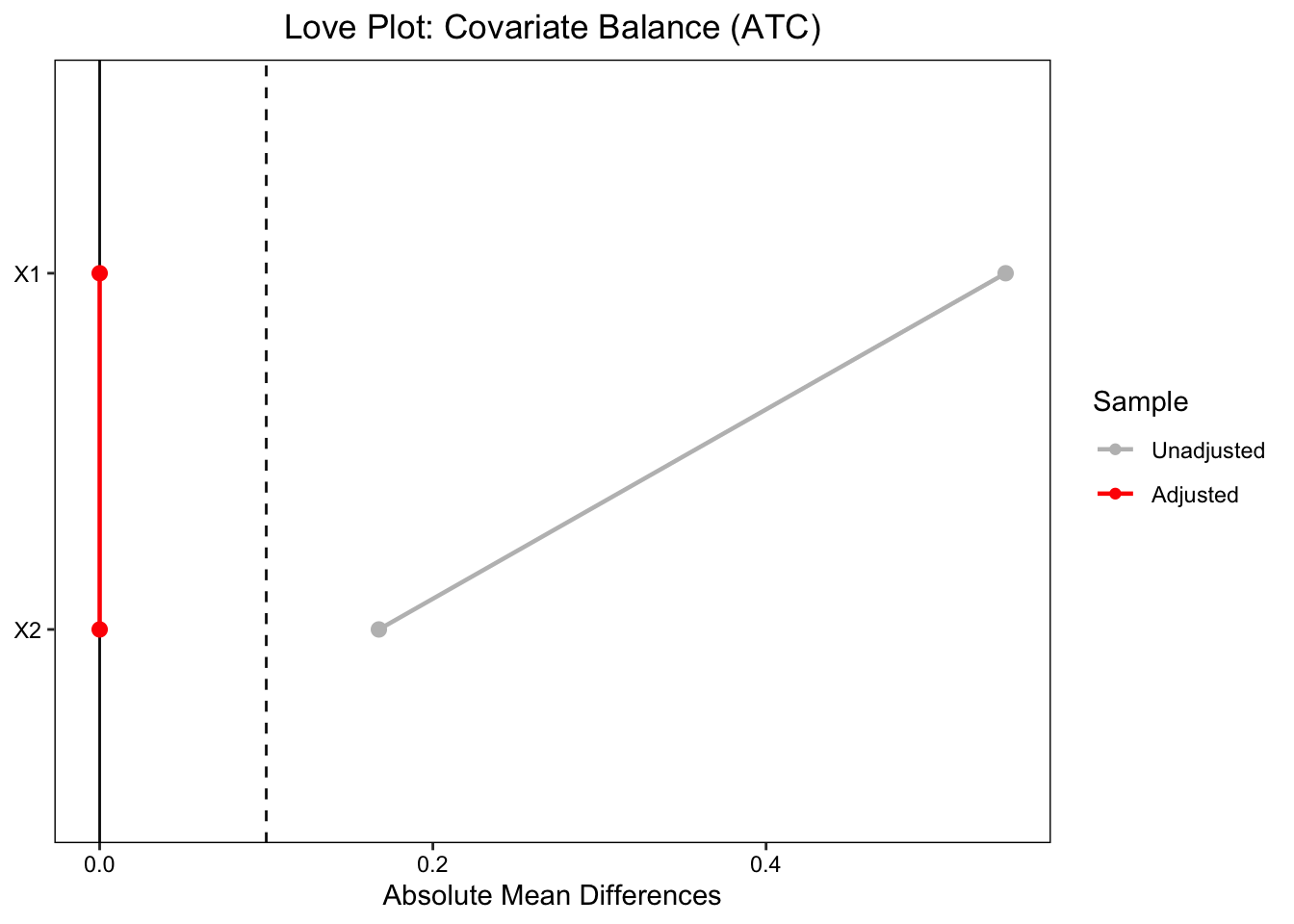
# ATC love.plot (w_atc,stat = "mean.diffs" , abs = TRUE , thresholds = c (m = .1 ),var.order = "unadjusted" , line = TRUE ,title = "Love Plot: Covariate Balance (ATC)" ,colors = c ("grey" , "red" ))
Warning: Standardized mean differences and raw mean differences are present in
the same plot. Use the `stars` argument to distinguish between them and
appropriately label the x-axis. See `?love.plot` for details.
DiD
エントロピーバランシングによる重み付けをしたうえで,DiDを実装してみる. 2 by 2で考えてみよう.
\[
Y_{it} = \alpha + \delta \cdot Post_t + \tau \cdot Treat_i + \beta \cdot(Post_t \times Treat_i) + \varepsilon_{it}
\]
# パッケージの読み込み library (ebal)library (survey)# 再現性確保 set.seed (123 )# サンプル数 <- 500 # 共変量(時間不変) <- rnorm (n)<- rbinom (n, 1 , 0.5 )<- runif (n)# 処置群かどうか(0: control, 1: treated) <- rbinom (n, 1 , 0.5 )# 時間(0: before, 1: after) <- rbinom (n, 1 , 0.5 )# 真のDID効果:treat × time の交差項 <- 2 # アウトカム生成:線形関数 + DID効果 + 誤差項 # 基礎回帰式: y = beta0 + beta1*treat + beta2*time + beta3*treat*time + e <- 1 + 0.5 * treatment + 1.5 * time + true_effect* treatment* time + 0.3 * X1 - 0.7 * X2 + 0.5 * X3 + rnorm (n)# データフレームにまとめる <- data.frame (X1, X2, X3, treatment, time, y)
# 共変量マトリクス <- as.matrix (df[, c ("X1" , "X2" , "X3" )])<- df$ treatment # 1 = treated, 0 = control # エントロピーバランシング:control を reweight <- ebalance (Treatment = Tr, X = X_covariates)
Converged within tolerance
# 重みを df に追加(treated=1には重み1, controlにはeb$w) $ w <- NA $ w[df$ treatment == 1 ] <- 1 $ w[df$ treatment == 0 ] <- eb$ w
# surveyパッケージのデザインオブジェクトを作成 <- svydesign (ids = ~ 1 , data = df, weights = ~ w)# 回帰モデル:DID形式(treatment × time 交差項) <- svyglm (y ~ treatment * time, design = design)# 結果の表示 summary (did_model)
Call:
svyglm(formula = y ~ treatment * time, design = design)
Survey design:
svydesign(ids = ~1, data = df, weights = ~w)
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 1.00294 0.09372 10.702 < 2e-16 ***
treatment 0.59123 0.14722 4.016 6.83e-05 ***
time 1.32843 0.13062 10.170 < 2e-16 ***
treatment:time 2.08285 0.19791 10.524 < 2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for gaussian family taken to be 1.211858)
Number of Fisher Scoring iterations: 2
正しい処置効果が推定されている.